(R) drakeでkeras mnistを動かしてみて動いたので記録。
チュートリアルをコピペ・整形したら出来るので本文は特に読まなくて良いですよ
RStudioのkerasパッケージのREADMEにある、mnistのサンプルを keras.rstudio.com
drakeパッケージを使って動かしてみた、というだけの記録。
drakeパッケージはCRAN版のバージョンが上がって、依存パッケージが変わってます。
上記drakeパッケージのGitHubリポジトリのInstallationの章を参照して、Bioconductorからgraphパッケージをインストールしてください。指示に従えば特に困ることはないはず、です。
ちょっと前に書いた記事の内容が、CRAN版でも必要になった、ということですね。
drakeパッケージは日々活発に開発がされていますので、GitHub版を追いかけるのもよいかもしれません。 まぁ、もうちょっと安定するまで触らなくてもよいのでは…げふんげふん
コード
貼る意味あるんか…
plan <- drake_plan ( mnist = dataset_mnist(), x_train_ = mnist$train$x, y_train_ = mnist$train$y, x_test_ = mnist$test$x, y_test_ = mnist$test$y, # reshape & rescale x_train = array_reshape(x_train_, c(nrow(x_train_), 784))/255, x_test = array_reshape(x_test_, c(nrow(x_test_), 784))/255, y_train = to_categorical(y_train_, 10), y_test = to_categorical(y_test_, 10), model = keras_model_sequential() %>% layer_dense(units = 256, activation = 'relu', input_shape = c(784)) %>% layer_dropout(rate = 0.4) %>% layer_dense(units = 128, activation = 'relu') %>% layer_dropout(rate = 0.3) %>% layer_dense(units = 10, activation = 'softmax') %>% compile( loss = 'categorical_crossentropy', optimizer = optimizer_rmsprop(), metrics = c('accuracy')), history = model %>% fit(x_train, y_train, epochs = 30, batch_size = 128, validation_split = 0.2 ) )
でplanを作って、下記で可視化と実行、結果の確認。
config <- drake_config(plan) vis_drake_graph(config) make(plan) loadd(history) plot(history)
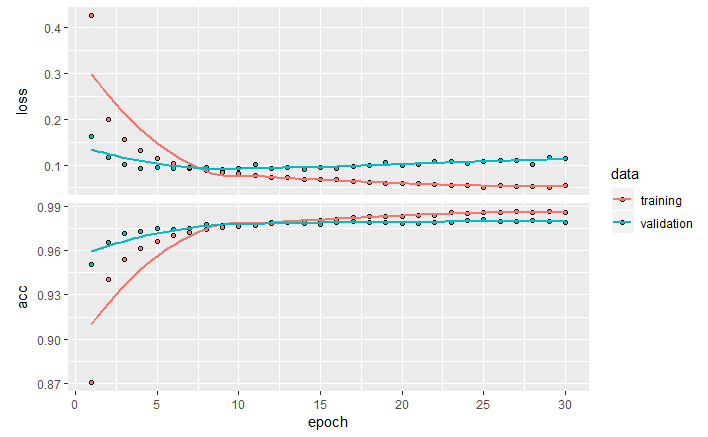
という感じでまぁ動いてよかったよかった…(?)
まぁ今更ですが、drake_plan()の中では<-ではなく=を使う。同じ変数を左辺に2度置けない。kerasのようにlayerをいじっていけるような場合(再代入不要な場合)はそのまま書いても大丈夫そう?です。
なお、書いたら動いた、って感じなので、細かいところは追えていません。
drakeパッケージもいつの間にかREADMEがすごい成長していて、そろそろ読み直さないと…
しかし、挙動が変…。
キャッシュ消してしまえばよいのですが、どうも挙動が変な気が…。
先のconfigを一度見てしまうと、plot()できなくなります(再度loadd()すればよいのだが…)
GitHub版では解決してるかもしれないし、こちらの環境かもしれないのですが。
いやまぁ、そもそもkerasの結果をdrakeで管理したいですかね?どうですかね…
ということで、やってみた系の記事でした。mnistは軽いので適当に実行して結果見るのに良いですね。
snakemakeも見てみようか…
RからPython実行すればよいかーと思っていましたが、その場合はsnakemakeパッケージをPythonで使えばよさそう?(と、drakeのREADMEに書いてある)
Snakemake — Snakemake 5.1.5+0.gb3233c8.dirty documentation
が、reticulateパッケージを介してRからPythonを動かせるので、これをやりたい。多分やればできるのでそのうちやる…。
と、いうことで・・・
Enjoy!!