最後まで行ってなかったので。
この記事の続きです。 niszet.hatenablog.com
注意点
以下、とりあえずやれるところまでやってみたものの結果、よくわかっていませぬ。
コードはコピペしたら動きますが、それ以上の情報はここにはないのでご容赦ください。
つづき
ホクソエム親分の資料、
のExercise 3です。
直前のコードのコピペだとこうなります
library(changepoint) data("HC1") data <- HC1 model <- cpt.meanvar(data, method= "PELT", test.stat="Exponential") cpts(model) #> integer(0) param.est(model) #> $rate #> [1] 0.0008198639 plot(model, cpt.width=3)
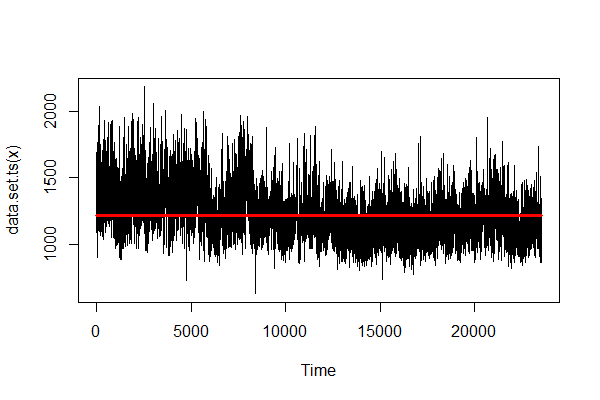
cptsの結果から分かる通り、全然推定してくれません。まぁ、そもそもExponentialに見えませんしね…
分布はNormal, Gamma, Poisson, Exponentialしか指定できないので、せっかくだから全部やってみます。
- Gamma
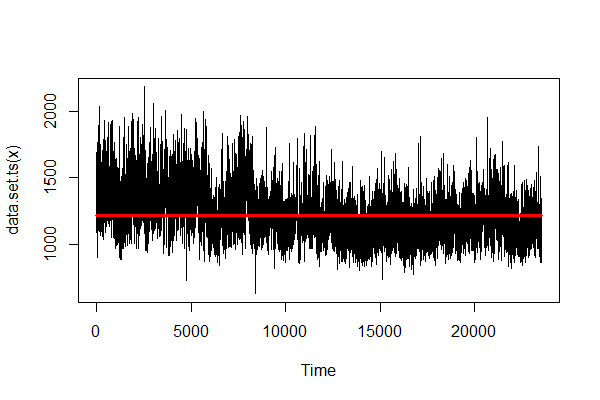
こちらも全然ダメ。
- Poisson
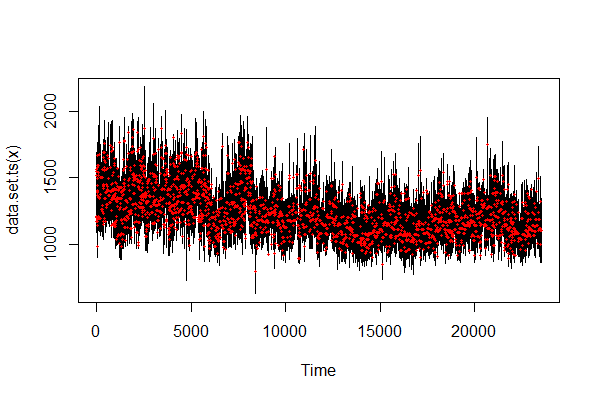 お。出ます…が、点が多すぎて気持ち悪いですね…
お。出ます…が、点が多すぎて気持ち悪いですね…
ちなみに、
length(cpts(model)) #> [1] 1760 length(data) #> [1] 23553
- Normal
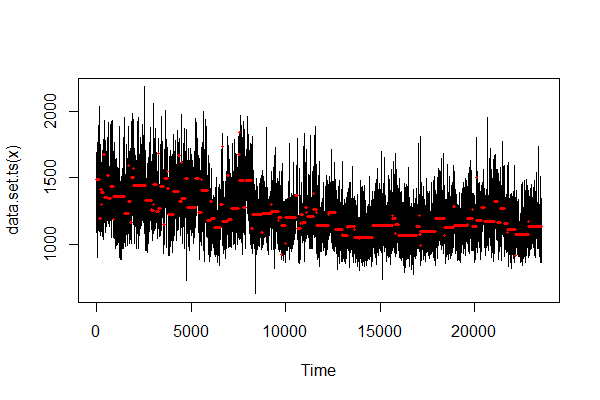
こっちなら変化点少なく出てくれますね。
length(cpts(model)) #> [1] 129
どれが正しいの?
どれでしょう…?
1番染色体のGC含有量…と言われてもピンと来ず…。
…が、含有量を時間に対してプロットしているので、今の値は直前の値に引っ張られているはずなので…つまりランダムウォークなのでは、と。 差分をとってプロット。
data <- data[2:length(data)]-data[1:length(data)-1] plot(data,type="l")
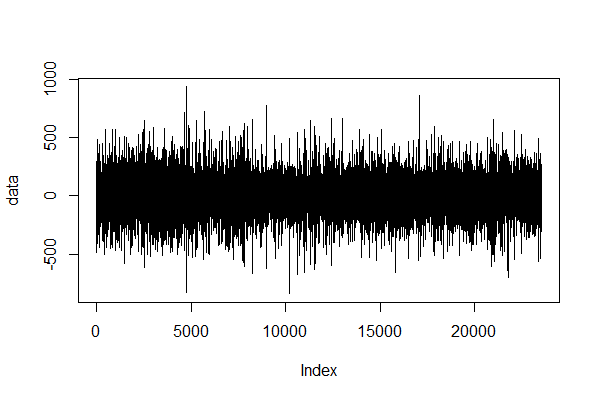
でも、これをmeanvarにかけると
model <- cpt.meanvar(data, method= "PELT", test.stat="Normal") cpts(model) #> [1] 7926 7928 param.est(model) #> $mean #> [1] 0.01400454 7.00000000 -0.03321813 #> $variance #> [1] 21828.13 0.00 16962.77
こんな感じに。平均7の分散0って何…。 このデータにコルモゴロフ-スミルノフ検定かけるとp<2.2e-16とか出てくるので正規分布ではないのかなー…
なお、Normal以外は負数があると使えないので差を取ったデータについてみてみるのはNormalのみです。
よくわからぬ…
とりあえずやってみたものの、今の自分では良く分かりませんでした…。いずれ解決させたいと思います。